Wurzelbiologie und Symbiose
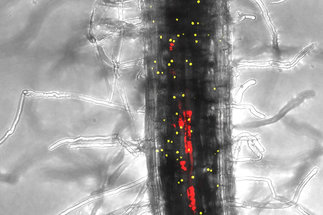
Die Abteilung von Prof. Dr. Caroline Gutjahr untersucht die Symbiose zwischen Bodenpilzen und Pflanzenwurzeln. Diese Symbiose wird Mykorrhiza genannt und ist von großer Bedeutung für das Wachstum und die Gesundheit von Pflanzen.
Zentrale Forschungsfragen sind, ob und wie die Etablierung der Symbiose von Umweltfaktoren beeinflusst wird und welche Moleküle dabei eine entscheidende Rolle spielen. Frau Gutjahr plant nicht nur die molekularen Vorgänge in der Pflanze zu untersuchen, sondern es sollen auch die bisher kaum untersuchten Pilze genauer analysiert werden. Des Weiteren beschäftigt sich die Abteilung mit der Frage, ob die molekularen Kenntnisse eingesetzt werden können, um Mykorrhiza-optimierte Nutzpflanzen zu züchten, welche zur Nachhaltigkeit in der Landwirtschaft beitragen könnten.
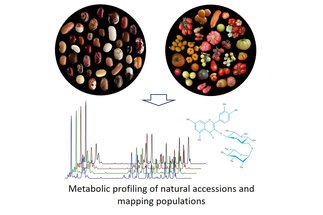
Weitere Forschungsthemen ihrer Abteilung sind Strukturforschung an Rezeptorproteinen, die bei Interaktionen der Pflanzen mit Mikroorganismen involviert sind (Alexander Förderer), sowie die Identifikation von Faktoren, die bei der Regulation des Primärstoffwechsels in photosynthetischen und heterotrophen Geweben eine Rolle spielen (AG Fernie). Darüber hinaus beschäftigt sich eine Projektgruppe (Saleh Alseekh) mit der Identifizierung von Genen, die an pflanzlichen Sekundärmetaboliten beteiligt sind.