Virusreplikation und Pflanzentoleranz
Viren können extreme Veränderungen in Zellen ihres Pflanzenwirts verursachen. Unser Ziel ist die von Pflanzenviren eingesetzten Mechanismen zu verstehen, die ein zelluläres Umfeld schaffen, welches für die Replikation ihres genetischen Materials förderlich ist, sowie die entsprechenden pflanzlichen Signalwege, die die Wirtszelle vor virusbedingten Schäden schützen zu entschlüsseln.
Viren kapern die Wirtsmembranen
RNA-Viren bilden Replikationskomplexe in verschiedenen Organellen ihrer Wirtszellen. Diese proviralen Strukturen trennen virale Faktoren und Wirtskomponenten vom Zytoplasma ab. Viele Studien sowohl bei Pflanzen als auch bei Tieren haben gezeigt, in welchem Ausmaß Viren die Wirtsmembranen während der Einrichtung ihrer viralen Fabriken umgestalten und wie allgegenwärtig diese Replikationsstrategie ist, was auf ein tief konserviertes Merkmal des viralen Lebenszyklus hindeutet. Trotz ihrer offensichtlichen Bedeutung ist wenig darüber bekannt, wie diese Strukturen gebildet werden. Wie konnte sich die virale RNA-Replikation so entwickeln, dass sie die Morphologie der Organellen so grundlegend verändern kann?
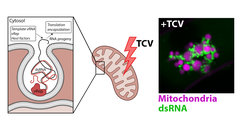
Rechts: Mit konfokaler Mikroskopie aufgenommene VRCs in lebenden, mit TCV infizierten Arabidopsis-Wurzeln. Mitochondrien sind magentafarben eingefärbt und dsRNA wird durch GFP-Fluoreszenz in Grün sichtbar gemacht.
Abgeändert aus: Clavel et Dagdas 2021 - Current Opinion in Plant Biology, 63:102106
Um diese Frage zu beantworten, setzen wir Proteomik, Biochemie, Zellbiologie und Genetik ein und untersuchen, wie virale Schlüsselproteine die Organellenmorphologie orchestrieren. Wir verfolgen einen vergleichenden Ansatz mit verschiedenen Pflanzenarten, um eine evolutionäre Perspektive zu gewinnen, wie diese Strukturen entstanden sind. Unsere Forschung hat das Potenzial, bisher unbekannte Aspekte des viralen Lebenszyklus zu identifizieren, die für den Anbau virusresistenter Nutzpflanzen genutzt werden könnten.
Zu unseren wichtigsten Forschungsfragen gehören unter anderem:
- Welches sind die konservierten Wirtsfaktoren, die für den Aufbau einer Replikationsorganelle notwendig und ausreichend sind?
- Wie orchestrieren virale Faktoren den Aufbau einer Replikationsorganelle?
- Bauen verschiedene Viren unterschiedliche Replikationskomplexe zusammen? Führen umgekehrt unterschiedliche Organellen zu unterschiedlichen Replikationskomplexen?
Pflanzen erkennen und beheben virenbedingte Organellenschäden

Wir haben vor kurzem einen Mechanismus entdeckt, den Pflanzen nutzen, um mit Schäden umzugehen, die durch die virale Zweckentfremdung ihrer Organellenmembranen verursacht werden. Bei einer Infektion mit verschiedenen Viren setzen Arabidopsis Pflanzen einen neu entdeckten selektiven Autophagie-Weg ein, der toxisches Material abbaut, das sich sonst im Zytosol ansammelt. Wenn die Autophagie nicht funktioniert, zeigen infizierte Pflanzen viel schwerere Symptome und entwickeln spontan abgestorbene Läsionen. Wir haben also eine unerwartete, schützende Funktion der selektiven Autophagie aufgedeckt und möchten ihre molekulare Funktionsweise verstehen. Um dies zu erreichen, konzentrieren wir uns auf neu identifizierte molekulare Akteure, die diesen Weg definieren und die Lücke zwischen toxischer Fracht und der Autophagie-Maschinerie überbrücken.
Besonders spannende Fragen sind:
- Wie schalten diese neuartigen Proteine von ihrer normalen Funktion zu ihrer Autophagie-bezogenen Funktion um? Wodurch wird die Toxizität der Autophagie-Fracht während der Infektion bestimmt?
- Welche(s) Signal(e) ist/sind notwendig, um den Autophagie-Prozess in Gang zu setzen?
- Können wir Auslöser außerhalb der Infektion identifizieren, die auf diesen Signalweg angewiesen sind?
- Wie sieht die frühe, zellautonome Reaktion von Pflanzenzellen aus, wenn eine virale Replikation wahrgenommen wird?

