Neuer Ansatz verbessert Genauigkeit der Vorhersage agronomisch relevanter Merkmale
Gatersleben/ Potsdam - Fortschritte bei Phänotypisierungsplattformen mit Hochdurchsatz (HTP) in Verbindung mit Genotypisierungstechnologien haben die Züchtung von Sorten mit gewünschten Merkmalen auf der Grundlage genomischer Vorhersagen revolutioniert. Dennoch fehlt bisher noch das Verständnis für die Ausprägung mehrerer Merkmale zu verschiedenen Zeitpunkten während der gesamten Wachstumsphase der Pflanze. Ein Forschungsteam, dem u. a. auch das Leibniz-Institut für Pflanzengenetik (IPK) und Kulturpflanzenforschung, das Max-Planck-Institut für Molekulare Pflanzenphysiologie (MPI-MP) und die Universität Potsdam angehören, hat jetzt einen computergestützten Ansatz entwickelt, um dieses Problem zu lösen. Die Ergebnisse wurden in der Zeitschrift „Nature Plants“ veröffentlicht.
Das Phänom einer Pflanze umfasst die Gesamtheit ihrer Merkmale zu einem bestimmten Zeitpunkt. Er ist das Ergebnis von genetischen Faktoren und von Umweltbedingungen sowie ihrer Wechselwirkungen. Das Verständnis dafür, wie sich das Phänom einer Kulturpflanze im Laufe der Zeit verändert, kann helfen, einzelne Merkmale zu bestimmten Zeitpunkten in der Entwicklung der Pflanze vorherzusagen. Doch das ist nicht nur wegen der Abhängigkeiten zwischen einzelnen Merkmalen schwierig, sondern auch wegen der Unterschiede in der Art und Weise, wie sich die Phänome bestimmter Genotypen im Laufe des Lebenszyklus einer Pflanze verändern.
Der klassische Ansatz der genomischen Vorhersage (GP) bei Nutzpflanzen trainiert maschinelle Lernmodelle anhand von Daten zu Merkmalen, die in einer Population von Genotypen zu einem bestimmten Zeitpunkt auf Grundlage genetischer Marker gemessen wurden. Bestehende GP-Ansätze helfen jedoch noch nicht bei der Vorhersage der Dynamik mehrerer Merkmale, d.h. der Expression mehrerer Merkmale zu verschiedenen Zeitpunkten über die gesamte Wachstumsperiode der Pflanze.
Das Forschungsteam stellte dynamicGP vor, einen computergestützten Ansatz, der die Vorhersage der Merkmalsdynamik während der Entwicklung von Nutzpflanzen erleichtert, für die phänotypische Zeitreihenmessungen für mehrere Genotypen von HTP-Plattformen verfügbar sind. „Wir haben gezeigt, dass dynamicGP ein effizienter computergestützter Ansatz zur Vorhersage genotyp-spezifischer Dynamiken für mehrere Merkmale ist. Dies wird durch die Kombination von genomischer Vorhersage mit dynamischer Modenzerlegung (DMD) erreicht“, erklärt David Hobby, Forscher am MPI-MP und der Universität Potsdam und einer der Erstautoren der Studie.
Anhand genetischer Marker und Daten aus der Hochdurchsatz-Phänotypisierung einer mehrelterlichen fortgeschrittenen Kreuzungspopulation von Mais und eines Diversitätspanels von Arabidopsis thaliana konnten die Forscher zeigen, dass dynamicGP genauere Vorhersagen zu der Gesamtheit der Merkmale liefert als die bisherigen Ansätze. „Wir haben festgestellt, dass die Entwicklungsdynamik von Merkmalen, deren Erblichkeit im Laufe der Zeit weniger variiert, mit höherer Genauigkeit vorhergesagt werden kann. Das ermöglicht Aussagen über die Vorhersagbarkeit von Merkmalen über den gesamten Entwicklungsverlauf hinweg“, sagt Dr. Marc Heuermann, Forscher am IPK und ebenfalls einer der Erstautoren der Studie.
Daher ebnet dynamicGP den Weg für die Untersuchung und Integration der dynamischen Wechselwirkungen zwischen Genotyp und Phänotyp während der Pflanzenentwicklung. Das Ziel ist es, die Vorhersagegenauigkeit agronomisch relevanter Merkmale zu verbessern. Zukünftige Entwicklungen von dynamicGP können auf Erweiterungen von DMD aufbauen, um auch die Auswirkungen von Umweltfaktoren zu berücksichtigen. Dies wird weitere Verfeinerungen des Ansatzes ermöglichen. Dies kann sowohl einen großen Einfluss auf die Züchtung von Pflanzensorten haben, die an bestimmte Regionen angepasst sind, als auch die Präzisionslandwirtschaft verbessern.
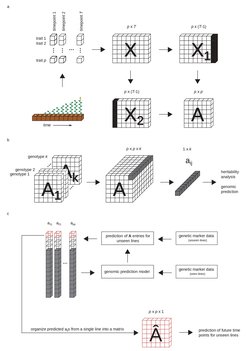
a) Daten zu phänotypischen Merkmalen (p) eines Genotyps werden über die Zeit (T) gesammelt und verwendet, um einen zeitinvarianten linearen Operator (A) zu finden, der das Phänom von einem Zeitpunkt zum nächsten transformiert. b) Wenn zeitaufgelöste Daten für mehrere (k) Genotypen verfügbar sind, erhalten wir einen p x p x k-Tensor, dessen Elemente bei Heritabilitätsanalysen und genomischen Vorhersagen als Merkmale behandelt werden können. c) Anschließend können Modelle für ungesehene Linien anhand ihrer genetischen Informationen trainiert und zur Vorhersage künftiger Zeitpunkte für die Linie verwendet werden.
Gemeinsame Pressemitteilung des IPK Leibniz-Instituts und des Max-Planck-Instituts für Molekulare Pflanzenphysiologie.
