"Wilde" Verwandte können Kulturpflanzen verbessern
Forschungsbericht (importiert) 2012 - Max-Planck-Institut für Molekulare Pflanzenphysiologie
Wildarten können verwendet werden, um die genetische Vielfalt von Kulturpflanzen zu erhöhen und ihren Wert für die landwirtschaftliche Produktion zu verbessern. Dafür ist ein umfassendes Verständnis der Stoffwechselwege und der Wechselwirkungen zwischen Genen, Erscheinungsbild (Phänotyp) und Umwelt notwendig. Das Metabolitenprofiling ist eine sich rasch verbreitende Technologie zur chemischen Phänotypisierung und diagnostischen Analyse von Pflanzen. Sie stellt einen leistungsstarken Ansatz dar, um die für die Stoffzusammensetzung ausschlaggebenden genetischen Faktoren zu bestimmen.
Verringerung der genetischen Vielfalt durch Auslese und Züchtung
Während der Domestikation unserer Kulturpflanzen wurden schwerpunktmäßig zwei Zuchtziele verfolgt: die Steigerung des Ertrages und die Beseitigung oder Verminderung unerwünschter Verbindungen in Pflanzen, wie z. B. von Bitterstoffen. Die Selektion auf diese beiden Zuchtziele erfolgte auf Kosten der genetischen Vielfalt von Kulturpflanzen. Mit Ausnahme von Mais besitzt die Mehrzahl unserer Nutzpflanzen schätzungsweise nur noch vier bis zehn Prozent der genetischen (allelischen) Vielfalt, die noch bei ihren „wilden“ Stammeltern vorhanden war [1]. Diese Situation gab den Anstoß dazu, die natürliche Variationsbreite auf der Ebene der Inhaltsstoffe zu untersuchen – unter besonderer Berücksichtigung derjenigen Stoffe, die einen besonderen Einfluss auf den Geschmack, den Ernährungswert und die pflanzeneigene Stresstoleranz besitzen.
Übertragung von Erbgut verwandtschaftlich weit entfernter Wildtypen auf Kultursorten
Durch die Zusammenarbeit mit der Arbeitsgruppe um Dani Zamir von der Hebrew Universität in Jerusalem konnten für die Untersuchungen am MPI für molekulare Pflanzenphysiologie Tomatenlinien genutzt werden, die dort (departments.agri.huji.ac.il/horticulture/staff-eng/zamir.html) erzeugt worden waren. Entstanden waren diese Linien durch Kreuzung einer Tomatenwildart mit einer Kulturtomate. Bei der Wildart handelte es sich um die besonders stresstolerante Wildart Solanum pennellii, bei der Kultursorte um eine Tomate, die als verarbeitetes Produkt in Form von Ketchup, Tomaten- bzw. Pizzasauce auf den Markt kommt.
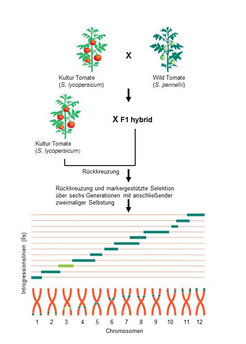
Abb. 1: "Bibliothek" der Introgressionslinien: Sie umfasst 76 Linien. Auf jedem der 12 Chromosomen der Elitesorte S. lycopersicum wurde ein Abschnitt durch Gene des Wildtyps S. pennellii ersetzt. Durch molekulargenetische Untersuchungen, gepaart mit Inhaltsstoffanalysen, können diejenigen Chromosomenabschnitte identifiziert werden, die für die Inhaltsstoffzusammensetzung der Tomaten verantwortlich sind.
Die aus der Kreuzung resultierenden Hybriden wurden mehrmals mit der Kultursorte rückgekreuzt, wodurch das Erbgut der Wildart immer weiter zurückgedrängt wurde. Zu guter Letzt wurden die Pflanzen geselbstet, das heißt sie befruchteten sich selber. Abbildung 1 zeigt eine schematische Darstellung der aus diesen Kreuzungen resultierenden „Bibliothek“ von Introgressionslinien.
Das Besondere dieser verschiedenen Introgressionslinien ist, dass sie jeweils einen unterschiedlichen Chromosomenabschnitt der Wildart besitzen, der an einer anderen Stelle auf den Chromosomen der Kultursorte lokalisiert ist. Solche Populationen haben drei große Vorteile: Auf den Chromosomen können relativ leicht die Regionen identifiziert werden, die für die phänotypische Varianz verantwortlich sind, das Erbgut weit entfernter „wilder“ Verwandter kann eingeführt werden, ohne dass die Fruchtbarkeit der Nachkommen darunter leidet, und es gehen keine erwünschten genetischen Phänotypen verloren.
Auffindung von Genorten, die ausschlaggebend sind für die chemische Zusammensetzung
Für diese Introgressionslinien wurde am Potsdamer MPI die Inhaltsstoffzusammensetzung reifer Früchte bestimmt und gemessen, um dann durch einen sog. QTL-Ansatz die genetischen Faktoren aufzuklären, die die Zusammensetzung der Inhaltsstoffe beeinflussen. QTL bedeutet Quantitative Trait Locus und bezeichnet einen Genort oder Genregionen, die auf die Ausprägung eines quantitativen Merkmals wie beispielsweise Wuchshöhe, Ertrag oder Inhaltsstoffzusammensetzung Einfluss nehmen. Bereits 2004 konnten Fridman et al. [2] durch einen solchen Ansatz den QTL für den Zuckergehalt auf ein einziges Nukleotid einengen. Darüber hinaus steht bei Tomatensorten, die für die Verarbeitung zu Ketchup und Soßen genutzt werden, der Zuckergehalt in direkter Beziehung zum Ertrag. In Anlehnung an diese Studie kam es bei der Untersuchung der oben beschriebenen Introgressionslinien zur Anwendung einer am Max-Planck-Institut für molekulare Pflanzenphysiologie entwickelten Methode, die eine viel umfangreichere Inhaltsstoffbestimmung ermöglicht.
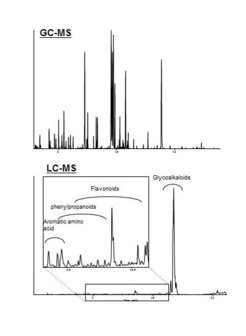
Um QTL – quantitative Genregionen – zu finden, bedarf es einer Reihe molekulargenetischer Analysen. Durch den Vergleich der genetischen Ausstattung der Introgressionslinien untereinander und mit der Kulturtomate sowie der Varianz ihrer Inhaltsstoffzusammensetzung können diejenigen Genregionen identifiziert werden, die für die betrachteten Inhaltsstoffe ausschlaggebend sind. Dadurch konnten in kürzester Zeit mehr als 1.000 die Inhaltsstoffe betreffende, sogenannte metabolomic Quantitative Trait Loci (mQTL) identifiziert werden. Eine schematische Darstellung derjenigen Stoffe, auf die die Untersuchungen abzielten, zeigt Abbildung 2. Durch die Nutzung von Hochdurchsatzverfahren konnte das Vorhandensein und der Gehalt von mehr als 400 Inhaltsstoffen ermittelt werden, inklusive der in Tomaten traditionell gemessenen Inhaltsstoffe wie Zucker, organische Säuren und leicht flüchtige Stoffe sowie Aminosäuren, Phenylpropanoide, Farbstoffe und Vitamine.
Besseres Verständnis der Pflanzenphysiologie durch Netzwerkanalysen
Die Auswertung der Ergebnisse durch hochkomplexe statistische Verfahren (Netzwerkanalysen) mit nachfolgenden physiologischen Analysen erlaubten mehrere wichtige Rückschlüsse auf die Physiologie von Tomaten. So wird zum einen sowohl der physiologische als auch der agronomische Ertrag weitgehend durch die Fotosynthese und den Transport der Assimilate in die Frucht bestimmt, zum anderen wirkt das den Samen umgebende Fruchtgewebe sehr stark auf die Inhaltsstoffzusammensetzung der Samen ein [3]. Beim Anbau der verschiedenen Tomatenlinien zeigte sich, dass die Inhaltsstoffzusammensetzung zu einem großen Anteil vererbt und weniger durch die Umwelt beeinflusst wird und dass heterozygote Linien, die also ein Allel der Kultursorte und eins der Wildsorte besitzen, mehr Inhaltstoffe besitzen als diejenigen Linien, die homozygot für die Introgression sind, ohne dass die Erträge darunter leiden. Angesichts der Dominanz des Einsatzes von Hybriden in der Landwirtschaft ist diese Erkenntnis besonders wichtig, da dies die Möglichkeit des Metabolomics-assisted breeding eröffnet: Die Inhaltsstoffzusammensetzung wird in den Züchtungsprozess einbezogen [1], [4].
Verbesserung des Nährwertes und des Geschmacks von Nutzpflanzen
Im Rahmen der Grundlagenforschung der letzten Jahre konnten mehrere der oben beschriebenen Effekte genetisch erklärt werden. Besondere Aufmerksamkeit wurde dabei wichtigen ernährungsphysiologischen Bestandteilen wie Vitaminen und essenziellen Aminosäuren gewidmet ([5], [6]) und flüchtigen Komponenten, die zu den geschmacksgebenden Inhaltsstoffen gehören [7], [8], Tabelle 1.
Aktuelle interdisziplinäre Forschung, die Pflanzengenetiker und Biochemiker genauso einbezieht wie Genom-Sequenzierer, Ernährungsexperten und Mediziner, wird eine Ära ermöglichen, in der die Züchtung von Pflanzen zumindest teilweise auf der Basis ihrer chemischen Zusammensetzung erfolgen kann, was zu einer Verbesserung der Ertragssicherheit, des Geschmacks oder des Nährwertes beitragen wird. Das große Potenzial dieses Ansatzes wird deutlich, wenn man die weiterhin wachsende Weltbevölkerung, Umweltzerstörungen und die Konkurrenz zwischen dem Anbau von Nahrungspflanzen und Nicht-Nahrungspflanzen in Betracht zieht.

Tabelle 1 Liste der geschmacksbeeinflussenden flüchtigen Verbindungen und ihrer Vorstufen


